作者 | 论文团队
编辑 | ScienceAI
如同人类拥有语言,生命世界也有一套由氨基酸序列构成的「分子语言」—— 蛋白质 。近年来,人工智能领域的蛋白质语言模型(PLMs)展现出解码这套语言的强大能力,能够精准预测蛋白质的结构与功能。
然而,这些尖端模型的训练与使用,往往需要深厚的机器学习专业知识和编程能力,这在 AI 开发者与广大生物学家之间形成了一道鸿沟。
为了打破这一壁垒,西湖大学原发杰团队首先提出了一种新颖的蛋白质表征方法 —— 将蛋白质一维序列与三维结构相结合形成「结构感知」词汇表并据此训练出了蛋白质语言大模型 Saprot。
在此基础上,团队进一步推出了 SaprotHub 开源平台 。该平台旨在将 Saprot 等一系列先进蛋白质语言模型的能力开放给生命科学领域研究者,它也是开放蛋白质模型联盟(Open Protein Modeling Consortium, OPMC)为推动全球科研协作、共建开源社区而迈出的关键第一步。
这项研究成果已于近期发表在国际顶尖期刊《自然・生物技术》(Nature Biotechnology)上,论文题为《Democratizing Protein Language Model Training, Sharing and Collaboration》。

论文地址:https://www.nature.com/articles/s41587-025-02859-7
蛋白质研究的挑战:从模型「孤岛」到协作「蓝海」
蛋白质是生命活动的基石,而近年来,以 AlphaFold2 为代表的蛋白质语言模型(PLMs)在预测蛋白质结构与功能方面取得了革命性突破。然而,这些强大的 AI 工具如同精密的专业设备,其训练和部署通常需要深厚的机器学习知识,这为广大从事实验研究的生物学家设置了难以逾越的技术鸿沟。从复杂的编程环境配置,到海量数据的预处理,再到模型训练和评估,整个流程充满了挑战。这不仅限制了 AI 技术的普及,也减缓了其在医药、生物技术等领域的创新应用进程。
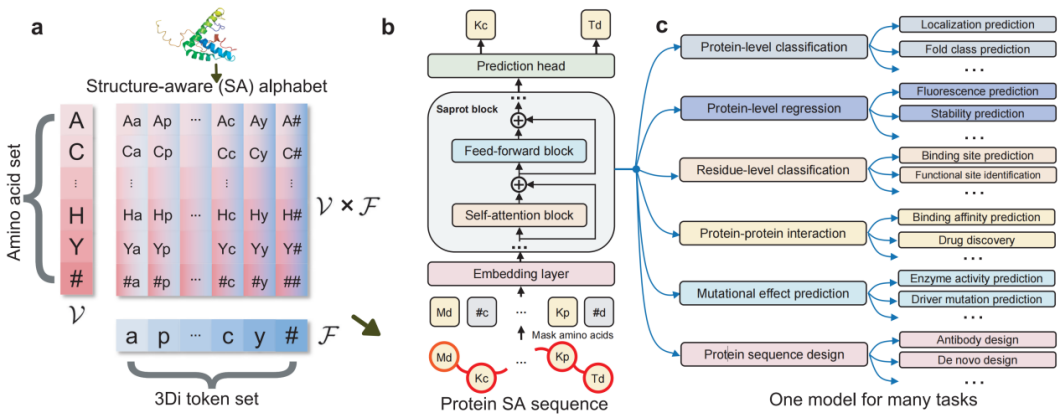
图 1. Saprot 模型架构
SaprotHub:三大支柱构建的开源协作新范式
为了应对这一挑战,团队构建了以 SaprotHub 为核心的一站式解决方案,它不仅是一个平台,更是一个融合了前沿 AI 大模型技术、开源工具和全球社区的完整生态系统:
核心引擎 — Saprot 语言模型:Saprot 是本项工作的基石。它开创性地提出了一种「结构感知」(Structure-Aware)词汇表,将蛋白质的一维氨基酸序列与其三维局部结构信息进行联合编码,从而构建出一种全新的蛋白质「语言」。同时,研究团队基于 AlphaFold2 预测的数千万个蛋白质结构,采用了 64 块 NVIDIA A100 GPU,经过数月训练完成了 Saprot 模型的训练。其性能在数十项蛋白质功能预测任务中得到了充分验证,并成功超越了如 ESM-2 等业界顶尖模型。自发布以来,Saprot 模型在学术界与工业界获得了广泛的关注和应用。相关论文被引用已超过 200 次,模型累计下载量逾 70 万次,并获得了大量来自社区的真实生物实验验证,彰显了其作为基础模型的影响力。值得一提的是,Saprot 于 2024 年 5 月登顶 ProteinGym 蛋白质突变效应预测排行榜,并在此后近半年的时间里持续排名第一。
开源工具 — 「一键式」蛋白质语言模型训练平台 ColabSaprot:为了将 Saprot 的能力释放给生命科学领域的研究者,团队基于免费的 Google Colab 云平台,通过数月开发,上万行的代码编写,实现了 ColabSaprot「一键式」开源训练平台。它将原本需要编写繁琐代码才能进行的蛋白质语言模型微调、功能预测等任务,简化为用户在网页上的几次鼠标点击,让不具备编程背景的生物学家也能轻松训练前沿蛋白质语言模型,实现从想法到验证的快速迭代 。为了方便研究者快速上手,团队录制了详细的教程视频,涵盖了从模型训练到使用等各个方面。
ColabSaprot链接:https://colab.research.google.com/github/westlake-repl/SaprotHub/blob/main/colab/SaprotHub_v2.ipynb
教程链接:
https://www.bilibili.com/video/BV1Y1i9YBEhv
https://www.youtube.com/watch?v=nmLtjlCI_7M
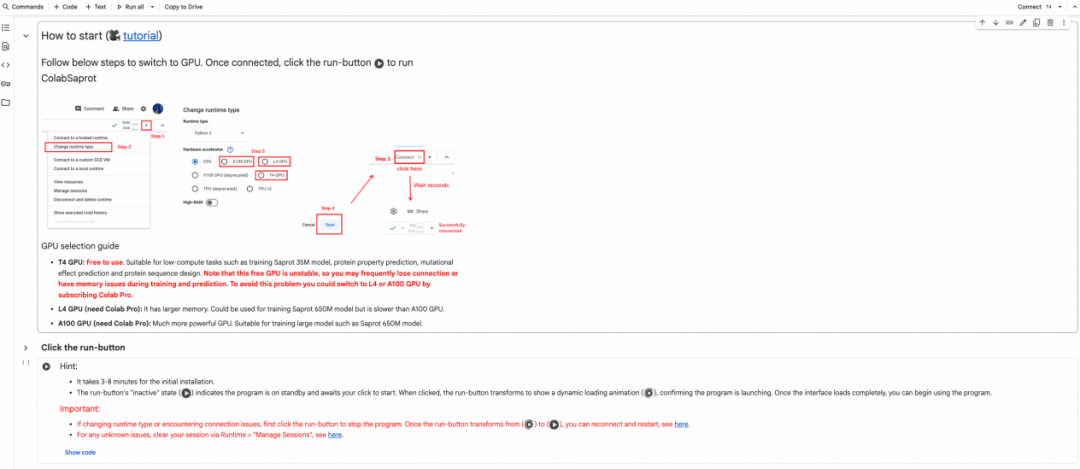
图 2. ColabSaprot 在线平台,具备细致的操作介绍以及简易的使用步骤
全球协作 — OPMC 成员共建的开放社区:SaprotHub 不仅仅是蛋白质语言模型的共享中心,更是开放蛋白质模型联盟(OPMC)理念的先行者。该联盟汇聚了来自西湖大学、麻省理工学院(MIT)、首尔大学、哈佛大学、慕尼黑工业大学、微软等全球数十家顶尖科研机构的研究力量 ,旨在共同推进蛋白质领域的蓬勃发展。为了实现开源共建的良性生态,团队采用低秩适应矩阵(LoRA)的方式保存模型权重,并建立了 SaprotHub 模型与数据仓库。通过将 ColabSaprot 开源平台与 SaprotHub 进行无缝耦合,OPMC 成员和全球研究者可以便捷地分享、下载和迭代模型。目前,SaprotHub 已经存储了数十种不同类型的蛋白质训练数据集以及可供研究者直接预测的蛋白质语言模型。为了方便研究者快速检索,团队针对性地开发了相应的搜索引擎,允许研究者根据关键词直接检索到相关的数据和模型。
SaprotHub 模型与数据仓库:https://huggingface.co/SaProtHub
搜索引擎:https://huggingface.co/spaces/SaProtHub/SaprotHub-search
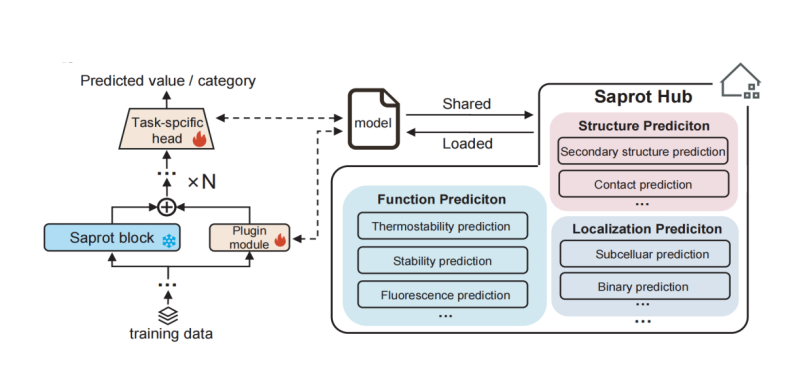
图 3. SaprotHub 利用 LoRA 技术存储模型权重,实现模型的便利共享
从虚拟到现实:计算机模拟性能验证与多项湿实验验证
SaprotHub 的价值不仅在于其便捷性,更在于其预测准确性。在团队开展的用户研究中,12 位没有 AI 背景的生物学研究者使用该平台,取得了与 AI 研究者相媲美的成果。
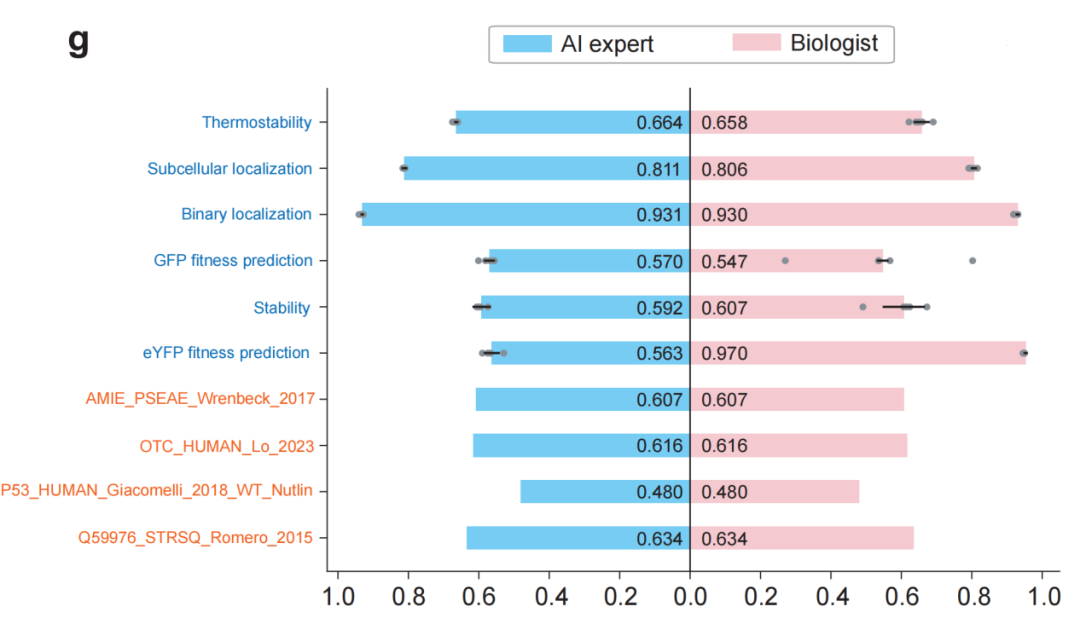
图 4. 生物研究者利用平台能够训练出和 AI 研究者相媲美的成果
更进一步,平台预测的有效性在一系列生物湿实验中得到了验证:
工业酶改造:一家生物技术公司利用 ColabSaprot 对一种工业用木聚糖酶进行改造,成功将酶的活性提升了 2.55 倍。
基因编辑工具优化:研究人员利用该平台对 TDG 基因编辑工具进行优化,预测出的多个新版本在实验中展现出翻倍的编辑效率。
荧光蛋白设计:平台还被用于设计更亮的绿色荧光蛋白(GFP),其中一个新设计的蛋白,其荧光亮度达到了原始版本的 8 倍以上。
这些成功案例证明,SaprotHub 能够将 AI 的预测能力转化为现实世界中的生物学功能突破。
核心突破
1. 全新蛋白质语言模型:发布了具备技术创新(结构感知词汇表)的 Saprot 蛋白质语言大模型。其在 14 项基准测试中性能超越了 ESM-2 等现有经典模型,已在该领域展现了其作为基础模型的影响力。
2. 开源协作范式:作为开放蛋白质模型联盟(OPMC)的第一步,汇聚了来自 MIT、哈佛、牛津、首尔大学等全球顶尖机构的智慧,为蛋白质领域建立了集模型训练、分享、合作、迭代于一体的开源社区平台。
3. 蛋白质语言模型技术民主化:通过「一键式」的 ColabSaprot 工具,将先进蛋白质语言模型的复杂训练和使用流程民主化,赋能全球不具备编程背景的生物学家,使其从 AI 的「使用者」转变为「创造者」和「贡献者」。
4. 真实场景验证:平台的有效性在工业酶改造、基因编辑工具优化等多个真实的湿实验场景中得到验证,展示了其通过计算机模拟辅助现实生物学突破的能力。
结语与展望
SaprotHub 的发布,不止是提供了一个工具。它以一个创新的自研蛋白质语言模型(Saprot)为基础,通过开源平台(ColabSaprot)来催化一个全球性的科研协作网络(OPMC)。这为 AI 辅助的生命科学研究提供了一种可持续发展的「开源、共建、共享」模式。目前,该生态已进一步集成了 ESM-2、ProtT5 等更多业界主流模型 ,开启了蛋白质科学的「大航海时代」。
西湖大学原发杰实验室现有 2026 年博士研究生招生名额,有意向者可将个人简历(含教育背景、科研经历、成果证明等)及相关材料投递至指定邮箱,邮件主题请注明「2026 博士申请 + 姓名」。
投递邮箱:[email protected]