编辑丨&
胚胎发育,一个生命形成的过程,从单个细胞逐渐分化,其中复杂的动态空间-时间表达决定了细胞命运决定和身体结构的形成。
但是在此过程中,传统成像方式却无法在时间上同时追踪多种波动的分子。固体胚胎成像提供了必要的灵敏度和容量,但缺乏时间分辨率。
上海交通大学提出了一种多尺度集成深度学习方法,以 1 分钟的分辨率从固定果蝇胚胎图像的核形态中精确推断绝对发育时间。
该研究以「Deep learning-based high-resolution time inference for deciphering dynamic gene regulation from fixed embryos」为题,于 2025 年 7 月 16 日刊登在《Nature Communications》。
论文链接:https://www.nature.com/articles/s41467-025-61907-7
解码胚胎发育
胚胎发育是由个体细胞中的复杂基因调控网络规划设计,如,在早期果蝇胚胎中,此时虽还未形成可见身体结构,但沿前-后(AP)轴的未来身体节段就已经被预先定义好了。
传统的实时成像受限于复杂的相互作用,固定成像的精细尺度又不足。深度学习的介入让这些问题变得逐渐清晰。该研究团队所采用的回归方法用于在 nc11-14 阶段推断早期果蝇胚胎的绝对发育时间,时间分辨率可达 0.3~1 分钟。
该回归方式采用了三种独立的卷积神经网络(CNN)模型进行集成学习,以此捕捉多尺度空间范围内的形态特征。它能够精确地从固定胚胎的标准 DNA 图像中推断出发育时间,不受品系的影响。
为了将发育时间与动态核信号相关联,团队收集了超过 30 个转基因果蝇胚胎的组蛋白 H2A-RFP 的时间序列图像,并观察它们各核周期之间核数量的显著差异。
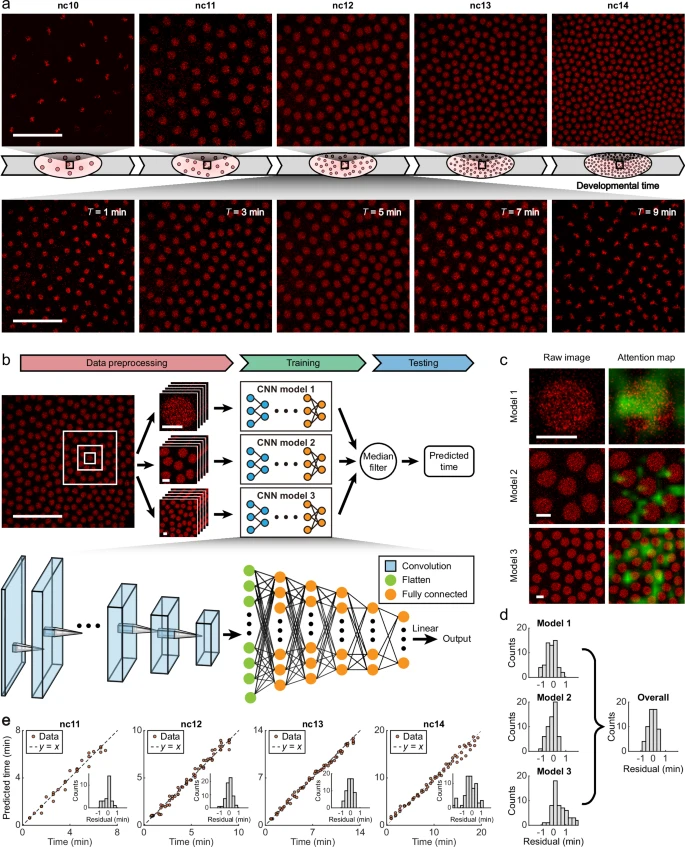
图 1:CNN 模型从活胚胎的核组蛋白图像预测发育时间。(图源:论文)
在前文中提到的三种独立 CNN 模型中,每种都有一个回归输出层,用于连续时间预测。这一点主要是通过每个胚胎图像分成多个不同大小的小窗口,捕捉不同空间尺度的时间依赖特征。
三个 CNN 独立打分,再采用中值滤波结合模型,干脆利落地得出结论。
与真实时间相比,模型的时间预测在所有核周期中既准确又精确,几乎没有偏差和变异性。几乎所有 nc11–14 的预测在 1 分钟内都是准确的,平均准确率达到了 96.25%。(nc11:100%,nc12:98%,nc13:100%,nc14:87%)
结果显示,基于 CNN 的方法在提取全面的图像特征进行精确时间推断方面具有极高价值,且团队还发现该方法对 nc14 的预测显著优于基于膜内陷的传统时间推断方法。
DNA 中的实验
核 DNA 相较于组蛋白,可以采用有机染料更方便的进行标记。但此处的难点是:DNA 图像和组蛋白图像长得不一样,模型不能直接搬家。
因此,实验里,先用 160 只固定胚同时为 DNA 和组蛋白染色,让 CNN 根据组蛋白推断胚胎的时间轴,并应用到 DNA 基模型上。适当校准图像与的对比度与饱和度后,这些 DNA 基模型的综合预测结果与基于组蛋白的时间推断结果高度一致。
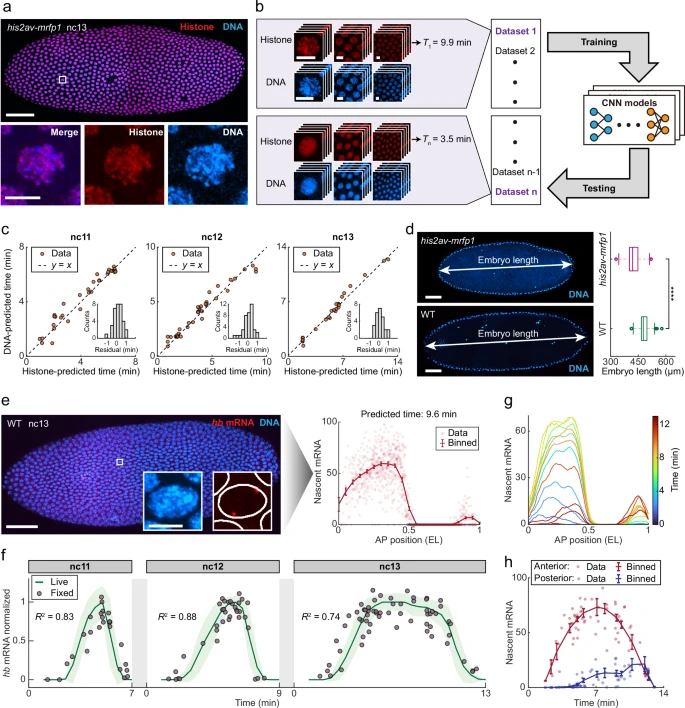
图 2:使用接力学习从固定胚胎的 DNA 图像中推断时间。(图源:论文)
如果想要将该方法推广到其他果蝇品系,特别是野生型(WT)的话,就需要对胚胎的 DNA 图像进行相应地缩放。
有了这个时间轴,对基因的工作才更好展开。
以一个多因素调控基因 Kr 为例。通过从 DNA 信号推断发育时间,团队迅速量化了固定胚胎在 nc11-13 期间核 Kr 转录和 Bcd 及 Hb 浓度的空间-时间分布。
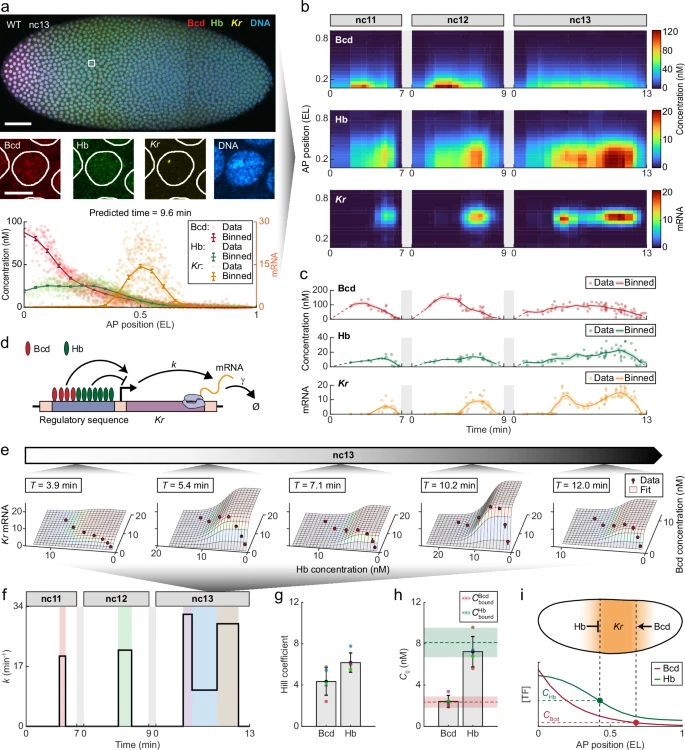
图 3:从固定胚胎中解析多个 TF 对 Kr 的动态调控。(图源:论文)
在 nc13 期间,Kr 出现了两个明显的表达峰值,而在之前这两个峰值是无法区分的,这一点凸显了新方法的敏感性。早期峰值的持续时间显著短于晚期峰值,解释了之前报道的 nc13 晚期 Kr 表达增加的现象。
小结
以早期果蝇胚胎为例,研究团队展示了他们的方法在单细胞和单分子水平上揭示关键发育基因的转录调控的强大能力。
与现有的基于 CNN 的胚胎分期方法相比,新方法更专注于快速变化的核信号,在精细化时间分辨力的同时,采用回归输出,允许连续时间观测。三个独立预测的网络覆盖了多个空间尺度,这使得模型有更强的稳健性与适应性。
放眼未来,这套框架可与单细胞测序、空间转录组、类器官成像无缝对接——从果蝇到斑马鱼,从小鼠胚胎到人类器官芯片,只需换一支染料,就能让所有「静态切片」秒变「高清延时影片」。
尽管眼下,这种方法还需大量胚胎成像,但它确实是一种有效通用的解决方案,可在不进行基因修饰的情况下准确量化发育动态。期待之后这种方法与高通量 RNA、蛋白质等的整合将会带来怎样的技术进步。



