作者 | 论文团队
编辑 | ScienceAI
药物 - 药物相互作用(DDIs)的准确预测对于保障药物安全和加速新药研发至关重要。然而,现有方法大多依赖整体分子结构或子结构表征,难以充分捕捉功能性 motif 之间的交互关系,而这恰恰是 DDIs 的根本原因。
与此同时,分子图像能够天然保留分子在二维 / 三维空间中的构象特征与视觉线索(如纹理、阴影、颜色和空间布局),这些信息往往难以通过单一图或序列表示捕捉,因而为刻画 motif 间的空间互作提供了独特优势。
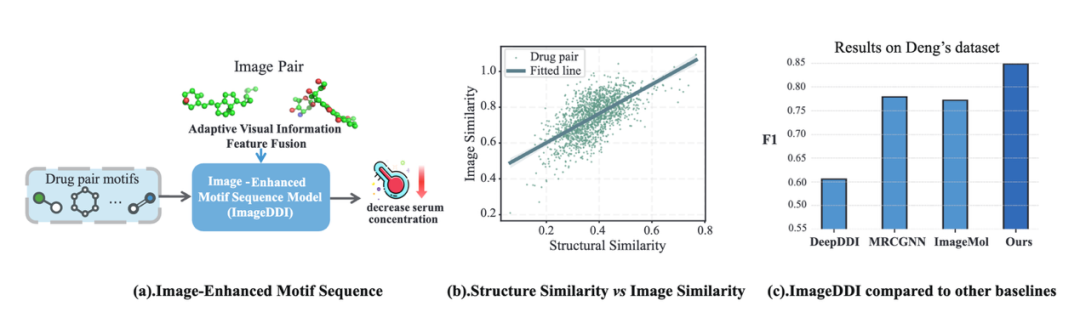
为此,湖南大学曾湘祥教授团队等提出 ImageDDI 框架:一种结合分子 motif 序列与分子图像信息的表征学习方法。
研究论文以《ImageDDI: Image-enhanced Molecular Motif Sequence Representation for Drug-Drug Interaction Prediction》为题发表在《Information Fusion》上。

论文链接:https://www.sciencedirect.com/science/article/abs/pii/S1566253525006463
代码链接:https://github.com/1hyq/ImageDDI
具体而言,ImageDDI 首先将药物切分为功能性 motif,并将药物对的 motif 序列输入基于 Transformer 的编码器,从局部结构层面建模。随后,引入分子全局图像特征,并通过自适应特征融合(Adaptive Feature Fusion) 动态整合局部与全局信息,从而获得更丰富、更具泛化性的药物表征。
方法
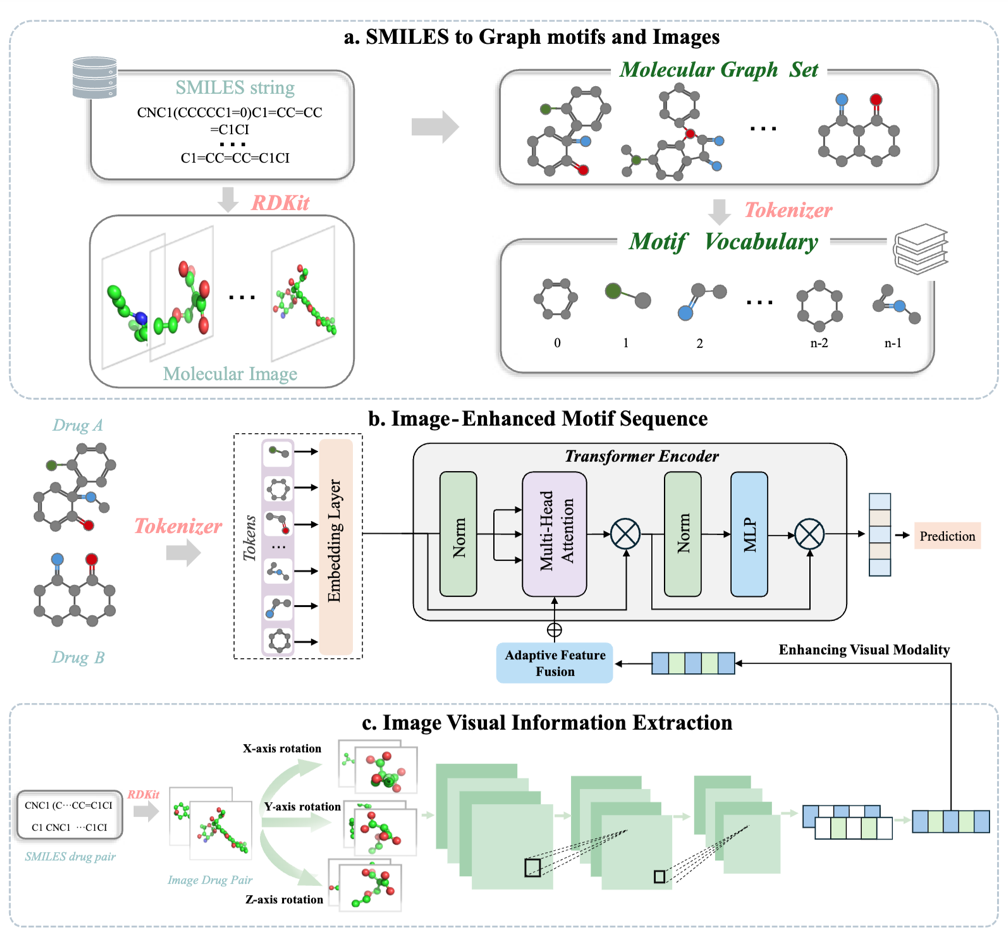
ImageDDI 方法整体框架概览:
(a) Motif 词表构建:首先,将分子按照 BRICS 规则切分为功能性 motif,并基于此构建 motif 词表,从而保证分子能够以更细粒度的子结构单元进行表征。
(b) 图像增强的 motif 序列建模:在得到药物对的 motif 序列后,将两个药物的 motif 序列拼接形成一个整体输入,并输入 Transformer 进行编码。在此过程中,通过引入 自适应特征融合(Adaptive Feature Fusion),将分子图像特征动态地融入注意力机制中,实现局部 motif 信息与全局视觉特征的联合建模。
(c) 分子图像特征提取:将分子在 X、Y 和 Z 三个轴上分别进行旋转,生成多角度视图,并提取其视觉特征。通过这一多视角的特征提取方式,模型能够学习到更加全面的分子空间结构信息,从而增强对 DDI 的预测性能。
(1) 从 SMILES 到图结构 motif 与分子图像
Motif 作为关键的局部结构,决定了分子在 DDI 中的行为,能够实现特定的结合与相互作用机制,从而影响特异性、亲和力以及整体预测效果。ImageDDI 将分子结构切分为 motif 并构建 motif 词表,使模型能够有效捕捉复杂的相互作用模式,从而提升 DDI 预测的准确性与可靠性。
具体来说,分子会被分解为功能性 motif,用于构建 motif 词表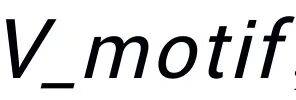 。这种方法能够保留关键信息,为下游任务提供坚实的基础,并提升分子表征的准确性。给定药物集合 D,每个药物
。这种方法能够保留关键信息,为下游任务提供坚实的基础,并提升分子表征的准确性。给定药物集合 D,每个药物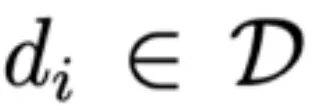 会通过 BRICS 方法处理,提取其 motif 集合
会通过 BRICS 方法处理,提取其 motif 集合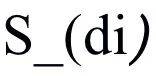 。BRICS 按照可切割键的规则对分子进行分解,新的
。BRICS 按照可切割键的规则对分子进行分解,新的 会被分配唯一 ID 并加入
会被分配唯一 ID 并加入 ,以确保下游分析的完整性与唯一性。
,以确保下游分析的完整性与唯一性。
二维图像(2D Image):使用 RDKit 的 Chem.Draw 模块将 SMILES 转换为标准化的二维拓扑分子图像,并统一原子 / 键的可视化方案和图像尺寸,以保证特征提取的一致性。
三维图像(3D Image):通过 RDKit 移除氢原子并采用 MMFFOptimizeMolecule () 与 MMFF94 力场优化生成分子构象(最多 5000 次迭代)。若优化不收敛,则迭代次数加倍并重复最多 10 次,若仍失败,则退回使用 2D 构象。
(2) 图像增强的 motif 序列建模
对于输入药物对 (dx, dy),首先提取其 motif 序列 Sdx 和 Sdy,并拼接为统一序列 S (dx,dy) = Sdx ⊕ Sdy。该序列既包含局部子结构特征,又建立了整体结构关系,为下游预测提供更丰富可靠的输入。为了有效建模 motif 序列并融合视觉信息,ImageDDI 采用 Transformer 结构,并在注意力机制中引入 自适应特征融合(Adaptive Feature Fusion),通过可学习的偏置项将视觉特征注入注意力公式:
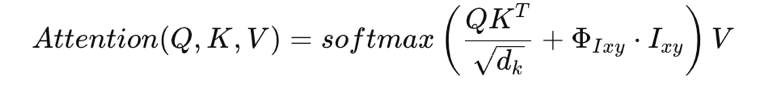
其中,Q、K、V 分别为 query、key、value 矩阵,ΦIxy 为基于药物对视觉特征 Ixy 的可学习偏置,用于动态调节视觉模态在特征融合中的贡献,从而优化视觉与结构特征之间的交互。多头注意力的输出进一步输入前馈网络(FFN),并结合残差连接与层归一化:
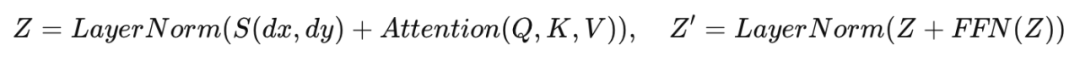
该方法扩展了 Transformer,使其能够同时处理 motif 序列和分子图像信息,从而更准确地建模分子间的相互作用。
(3) 分子图像特征提取
虽然 motif 能够有效捕捉局部结构信息,但不足以覆盖完整的分子交互模式。为此,ImageDDI 引入图像信息以学习全局交互特征。采用 ResNet18 作为编码 backbone,2D 编码器处理单帧图像,3D 编码器通过多视角均值池化获取特征。两个药物的视觉特征 Ix 和 Iy 拼接为联合表征 Ixy,用于后续预测。
实验
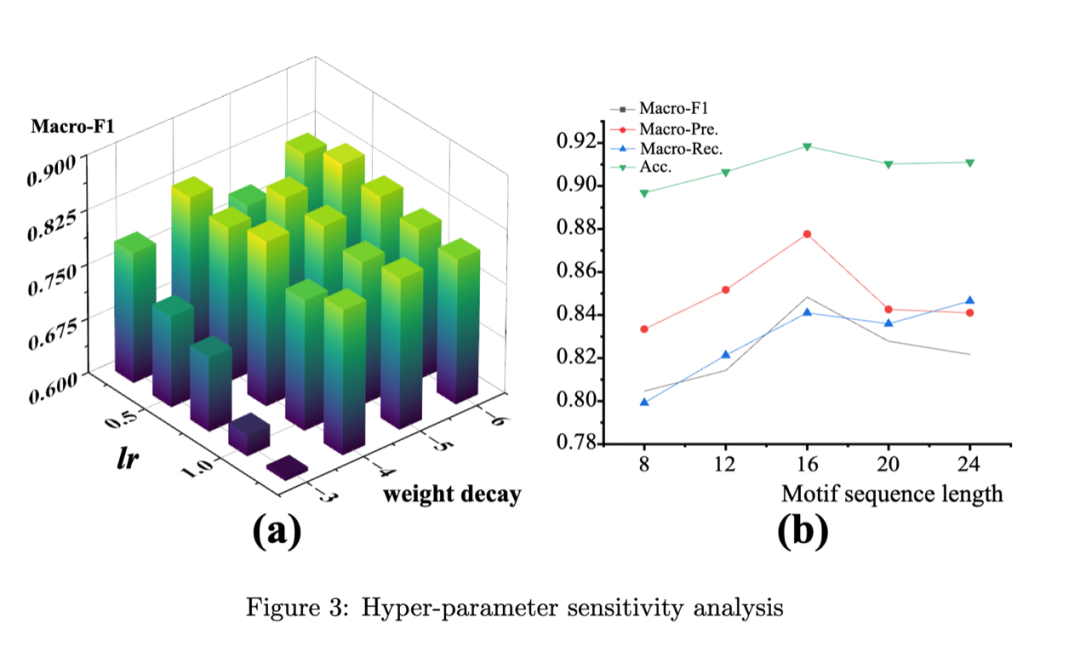
本节介绍了实验设置与结果。作者在三个常用数据集(Deng‘s dataset、Ryu’s dataset 和 DrugBank)上评估了 ImageDDI,在常规和归纳预测场景下均显著优于现有方法。随后,作者与多种代表性基线模型进行了比较,并通过消融实验验证了图像信息对模型性能的关键作用。进一步的超参数敏感性分析表明,合适的学习率、权重衰减和 motif 序列长度能够提升预测效果。最后,作者利用二维和三维可视化方法展示了 ImageDDI 对关键功能基团的准确捕捉能力,证明了模型的稳定性和可解释性。
常规场景:
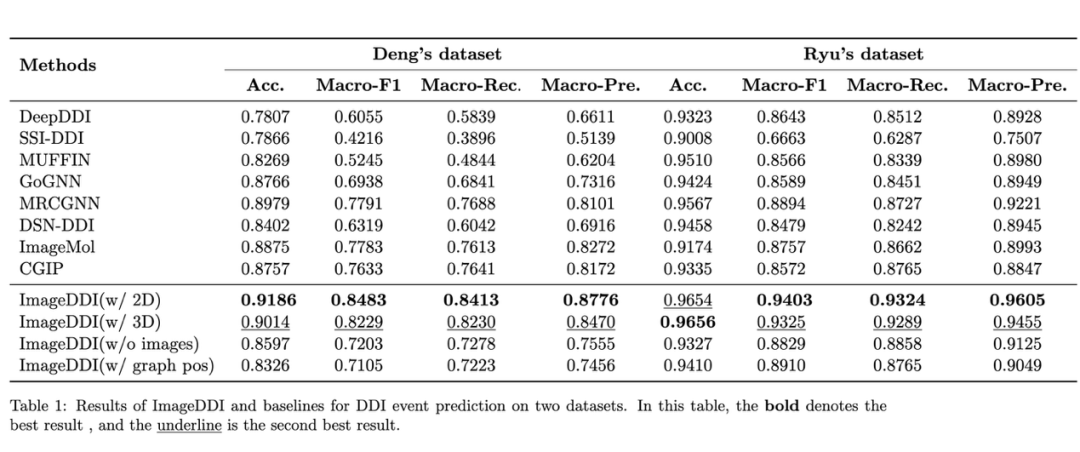
上图展示了 ImageDDI 与其他方法在 Deng’s dataset 和 Ryu’s dataset 上的表现。作者指出,ImageDDI 在 Accuracy 与 Macro-F1 上均显著优于现有方法,其中在 Deng’s dataset 上 Macro-F1 提升超过 10%,在 Ryu 数据集上提升超过 6%。虽然 3D 版本的表现略低于 2D 版本,但整体依然领先于所有基线模型,这说明图像增强的 motif 序列在表征局部与全局特征方面具有独特优势。
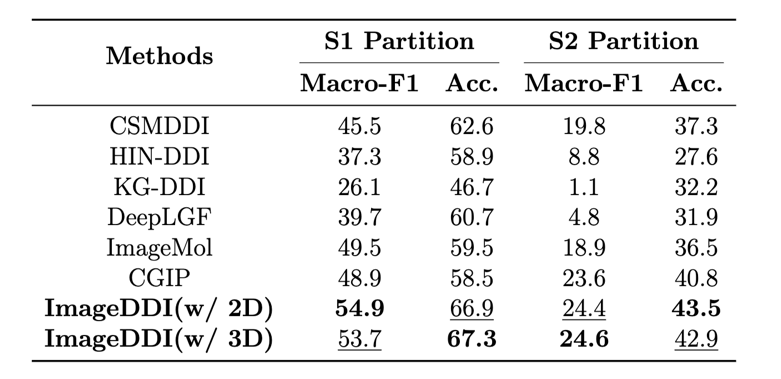
归纳预测场景:
为了进一步研究模型在 inductive 场景(冷启动环境) 下的能力,作者在 DrugBank 数据集上进行了药物级别的划分:将一部分在训练集中从未出现过的药物划为新药,用于测试模型对未知药物的预测能力。具体包括两种设置:在 S1 设置(新药 vs 已知药物) 下,ImageDDI 的 Macro-F1 和 Accuracy 分别比最佳基线提升 9.4% 和 7.4%;在 S2 设置(新药 vs 新药) 下,ImageDDI 同样优于现有方法 CGIP。这些结果表明,ImageDDI 在冷启动任务中同样表现突出,兼具鲁棒性和泛化能力,显著优于传统的图结构方法(如 MRCGNN)和图像方法(如 ImageMol)。
超参数实验:
视觉解释性案例研究:
2D 视觉可解释性:ImageDDI 能够通过注意力热力图准确识别药物对中的关键功能基团(motif),其结果与已知化学机制一致。这些结果表明,ImageDDI 能够准确识别关键 motif,即使在冷启动场景下,也能与化学领域对 DDI 的认识保持一致。
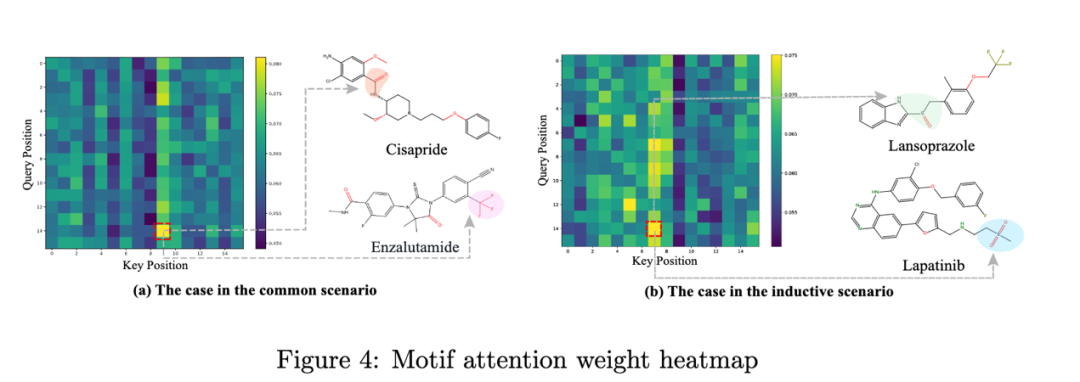
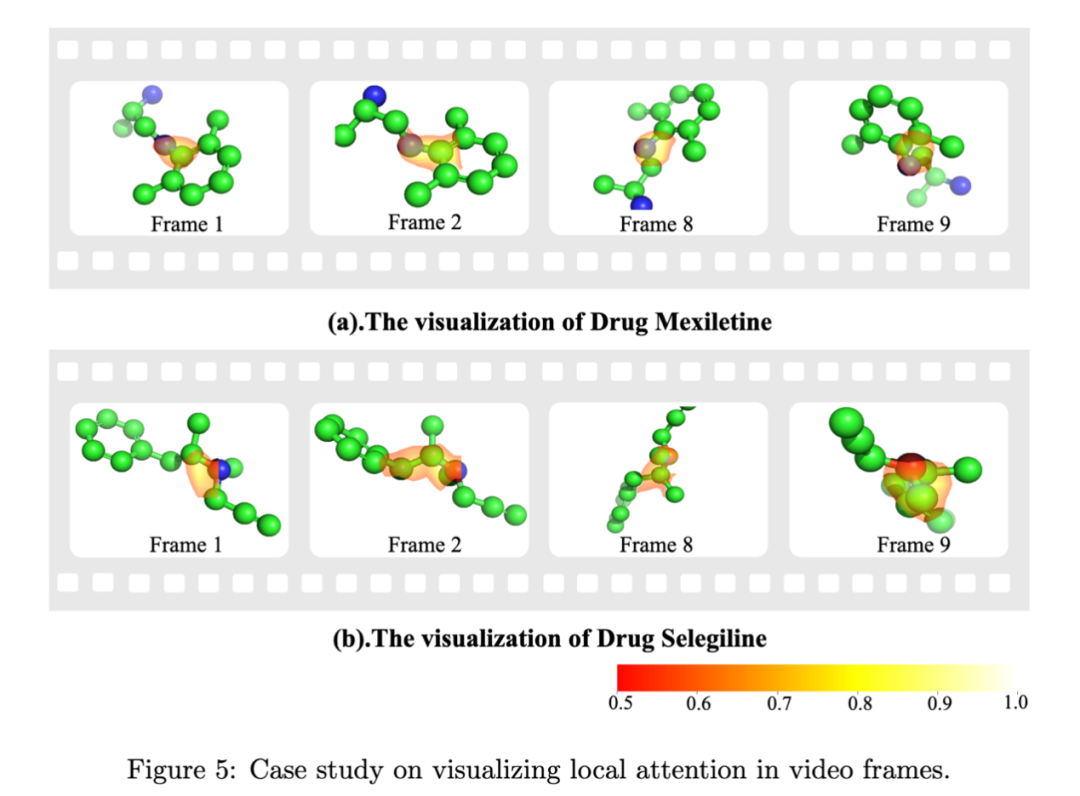
3D 视觉可解释性: ImageDDI 在不同视频帧下始终关注药物对中的相同关键结构基团,证明了其三维表征的稳定性与鲁棒性。
总结和讨论
在本研究中,作者首先分析了现有 DDI 预测方法在建模药物对 motif 交互上的局限性,指出仅依赖分子整体结构或子结构难以刻画相互作用的本质。为此,作者提出了一种图像增强的分子 motif 序列表征框架 ImageDDI,通过将分子切分为 motif 并序列化输入 Transformer,同时引入分子图像信息,并利用自适应特征融合(Adaptive Feature Fusion)联合建模局部 motif 与全局图像特征。
在实验中,ImageDDI 在常规预测和冷启动预测(S1:新药 vs 已知药物,S2:新药 vs 新药)两种场景下均显著优于基线方法。在 Deng‘s dataset、Ryu‘s dataset 和 DrugBank 数据集上,Macro-F1 和 Accuracy 提升明显,展现了强大的鲁棒性和泛化能力。同时,消融实验与可视化分析进一步验证了图像信息在捕捉关键功能基团中的重要作用。Grad-CAM 热力图显示模型在不同视角下始终聚焦相同 motif,体现了 ImageDDI 在空间感知与解释性上的优势。整体来看,ImageDDI 在性能、泛化性与可解释性上均取得突破,为 DDI 预测提供了新的思路。