编辑丨coisini
2024 年 5 月,Google DeepMind 发布了 AlphaFold3 模型,该模型能够对包括蛋白质、核酸、小分子、离子和修饰残基在内的复合物进行联合结构预测,堪称革命性模型。
但 Google DeepMind 没有立即公布其论文代码,而是在六个月后正式开源,允许学术研究者免费下载软件代码,但不允许将 AlphaFold3 用于商业应用。
非营利性人工智能研究联盟 OpenFold Consortium 一直致力于打造性能匹敌 AlphaFold3 的全开源结构预测基础模型。
现在,该研究联盟宣布发布 OpenFold3 预览版(OpenFold3-preview),这是一个开源深度学习模型,能够以高精度预测复杂蛋白质及其相互作用分子的三维结构。
开源地址:https://github.com/aqlaboratory/openfold-3
OpenFold3
OpenFold3 基于超大规模数据库进行训练,包含超过 30 万个公开可用的实验测定结构,以及超过 1300 万个 OpenFold 整理合成的结构。
OpenFold3 仅需氨基酸序列作为输入,即可预测蛋白质如何折叠成其三维结构。OpenFold3 新增了预测蛋白质与其他分子相互作用的能力,包括小分子配体、核酸等。
OpenFold3 基于 PyTorch 构建,并可在 NVIDIA NIM 上获取,这不仅实现了高速性能,还最大限度地减少了计算资源的使用。
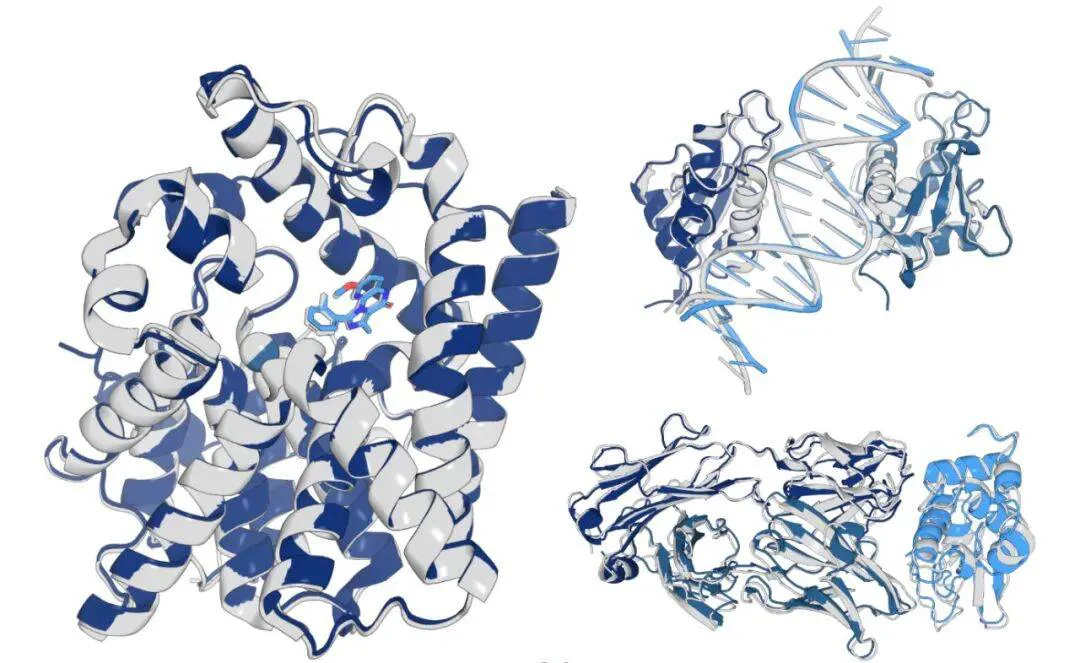
OpenFold3-preview 在生物分子结构预测方面的表现可媲美 SOTA 模型,并且是唯一一个在单体 RNA 结构预测中性能匹敌 AlphaFold3 的模型。
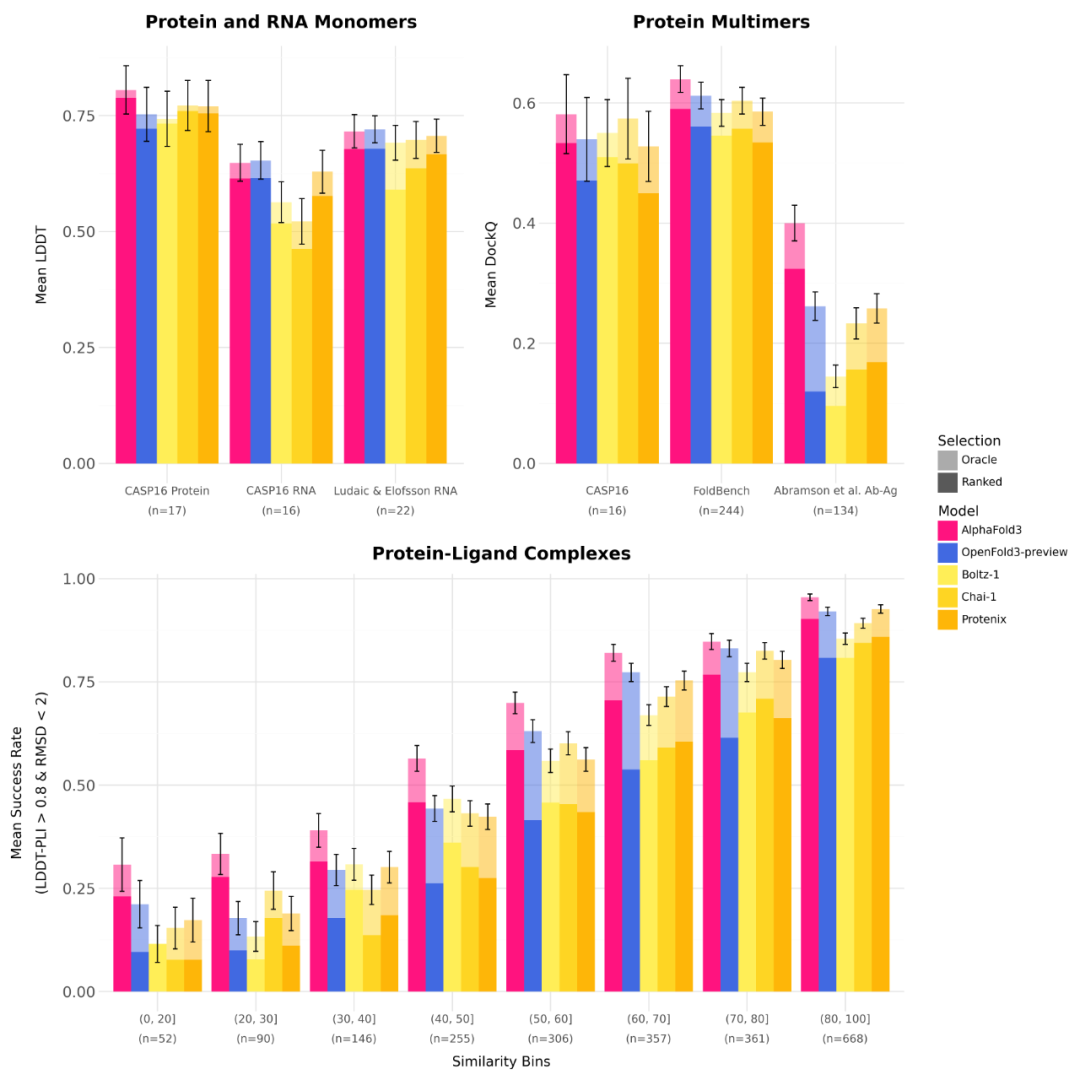
值得一提的是,OpenFold3 采用模块化设计,注重可用性,允许生物制药和材料科学公司修改模型,使其能够以原生格式解读数据,而不是让数据适应模型。OpenFold3 使生物分子建模变得可适应、可扩展。
可商用
相较于 AlphaFold3,OpenFold3 的一个关键优势在于其所有软件组件均采用 Apache 2.0 许可证,允许所有用户测试模型、使用新数据集进行训练、开发新应用并调整模型。
一些公司已计划利用 OpenFold3 加速新疗法和新产品的研发。知名制药公司诺和诺德将调整该模型以适应内部流程和专有数据,以支持新疗法的研发。细胞疗法公司 Outpace Bio 将使用该模型生成具有特定分子回路设计的新型细胞疗法。拜耳作物科学将应用 OpenFold3 研究来自植物、杂草和害虫的蛋白质,加速新型作物保护产品的研发。生物技术公司 Cyrus Biotechnology 将利用该模型设计用于治疗自身免疫性疾病的新型酶基药物。
此外,今年 6 月在英国启动的 OpenBind 计划将使用生成数据对 OpenFold3 进行微调。
OpenFold3 的全面开源与商用友好策略,将让结构预测模型可以在业界释放更多潜力。
参考内容:
https://www.nature.com/articles/d41586-025-03546-y
https://www.businesswire.com/news/home/20251028507233/en/OpenFold-Consortium-Releases-Preview-of-OpenFold3-An-Open-Source-Foundation-Model-for-Structure-Prediction-of-Proteins-Nucleic-Acids-and-Drugs