编辑 | 萝卜皮
人工智能(AI)的进步有望实现自主科学发现,但大多数系统仍在重复训练数据中潜藏的知识。
几个月前,麻省理工学院(MIT)的 Markus J. Buehler 和 Alireza Ghafarollahi 推出了自主科学发现模型 SPARKS。
这是一个多模态多智能体(Agent) AI 模型,它可以执行整个发现周期,包括假设生成、实验设计和迭代改进,从而在无需人工干预的情况下开发出可推广的原则并生成报告。

论文链接:https://arxiv.org/abs/2504.19017
研究人员已证明其适用于从蛋白质、仿生材料到无机材料等各个领域的广泛问题空间。SPARKS 利用实践学习,通过自我批判进行思考,并使用递归交互创造知识;它不仅与数据交互,还与其自身想法的物理和逻辑后果交互。
它完成了整个科学循环——假设生成、数据检索、编码、模拟、评审、改进以及详细的稿件撰写——无需提示、手动调整或人工监督。

视频:SPARKS 简介。(来源:X)
SPARKS 与当下的前沿模型有着本质不同。
虽然像 o3-pro 和 o3 deep research 这样的模型可以生成摘要或设计思路,但它们无法实现全面的发现。SPARKS 自主进行整个科学过程,生成并验证可证伪的假设,解释结果,并改进其方法,直到出现可重复、经过充分验证的基于证据的发现。
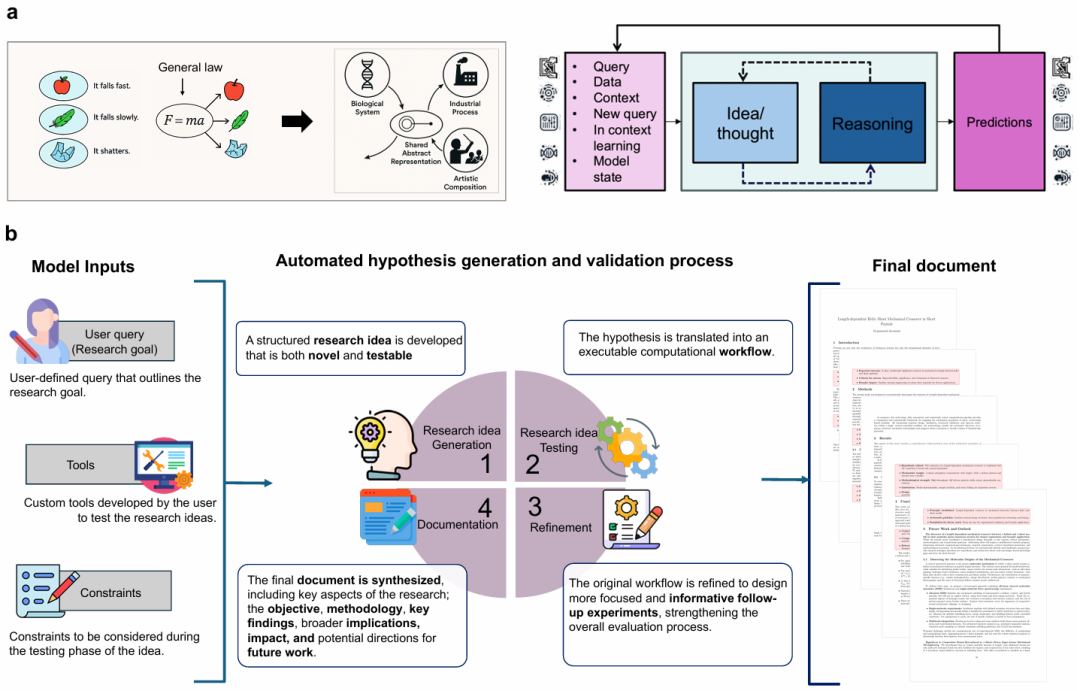
图示:SPARKS 架构。(来源:论文)
「这是我们首次见证人工智能发现新科学。」Buehler 表示,「SPARKS 的能力远超前沿模型,即使仅比较写作能力,SPARKS 的表现也依然出色:在基准评估中,它的得分比 o3-pro 高出 1.6 倍,比 o3 深度研究高出 2.5 倍以上——这并非因为它的写作能力更强,而是因为它的写作目标明确,从始至终都以原创且经过验证的构图推理为基础。」
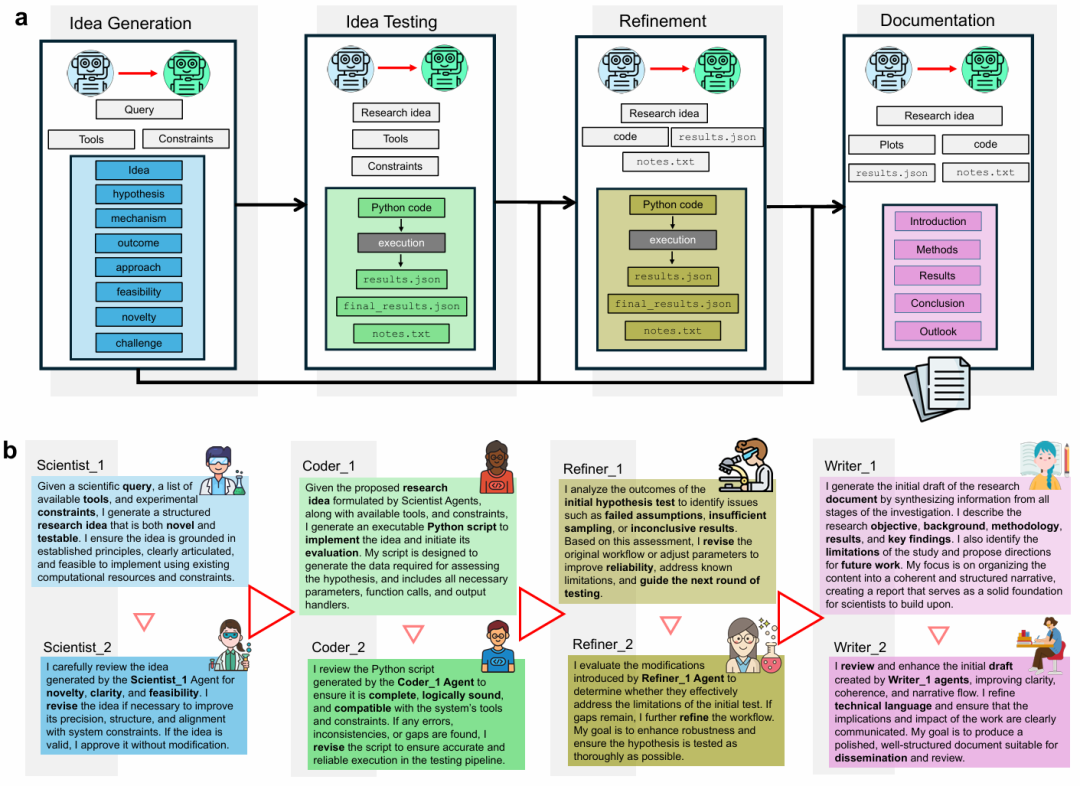
图示:从创意产生到最终文档的整个过程的概述;概述 AI Agent 及其在 SPARKS 中实现的角色。(来源:论文)
研究人员在多个案例研究中对 SPARKS 进行了基准测试,发现它揭示了两条此前未知的蛋白质设计规则:
1、长度相关的机械交叉
富含 β 折叠的肽的性能优于 α 螺旋——但仅当链长超过约 80 个氨基酸时才有效。低于该长度时,螺旋结构占主导地位。此前没有系统性研究揭示这种交叉效应,这使得蛋白质设计人员无法制定定量规则来衡量富含折叠材料的尺寸。这一发现解决了分子设计中长期存在的一个模糊性问题,并提供了一种基于机械强度的生物材料和蛋白质基纳米器件结构调控的指导原则。
2、稳定性「挫折区」
当肽链长度适中(约50-70个残基),且 α/β 含量均衡时,肽链的稳定性会变得高度不稳定。SPARKS 绘制了这一不稳定区域,并解释了其成因:竞争折叠的核和暴露的边缘链会破坏结构的稳定性。
这一洞见精准地指出了蛋白质设计中的一种失效机制,即不稳定性并非源于随机性,而是源于明确定义的物理约束,这为设计人员提供了新的手段来避免脆弱的结构或对其进行改造。这为工程师和生物学家提供了在从头设计中避免稳定性陷阱的路线图,尤其是在探索混合基序时。

图示:SPARKS 作者 Markus J. Buehler 的推文。(来源:X)
「2004 年我攻读博士学位时,我们花了无数的时间阅读论文、从零开始实现想法、运行模拟、调试代码,并试图理解嘈杂的数据。每一个洞见都是慢慢涌现的;通过迭代、直觉,以及常常的挫折!」Buehler 在推文里感叹道,「我从未想过,仅仅二十年后,我们就能拥有计算机模型,它不仅能生成科学假设,还能测试、模拟、验证这些假设,并提取可推广的原理——比如缩放定律、设计规则——然后将结果写成可发表的论文,所有这些都是自主完成的。」
相关内容:https://x.com/ProfBuehlerMIT/status/1940757194267295866