编辑丨coisini
化学键是指分子中相邻原子间的强烈相互作用。有限的原子种类和键合几何却能产生高度复杂的可设计结构,使大量原子能以精确定义的距离、取向和可预测的相互作用强度排列。
然而,由于蛋白质复杂的序列 - 结构关系,通过相互作用实现可预测键合来构建蛋白质组装体仍具挑战。
最近,华盛顿大学大卫・贝克(David Baker)团队提出了一种模块化蛋白质纳米材料设计策略,利用基于深度学习的生成工具设计蛋白质构建单元,结合规则配位几何结构与可定制的键合相互作用,使其能够通过简单几何原理指导组装多样化的封闭或开放结构。研究论文发表于《Nature Materials》上。

论文地址:https://www.nature.com/articles/s41563-025-02297-5
通用的蛋白质架构设计方法
该研究致力于开发一种通用的蛋白质架构(architecture)设计方法,通过构建具有特定配对界面的模块化结构单元,实现多样化三维结构的可编程组装。
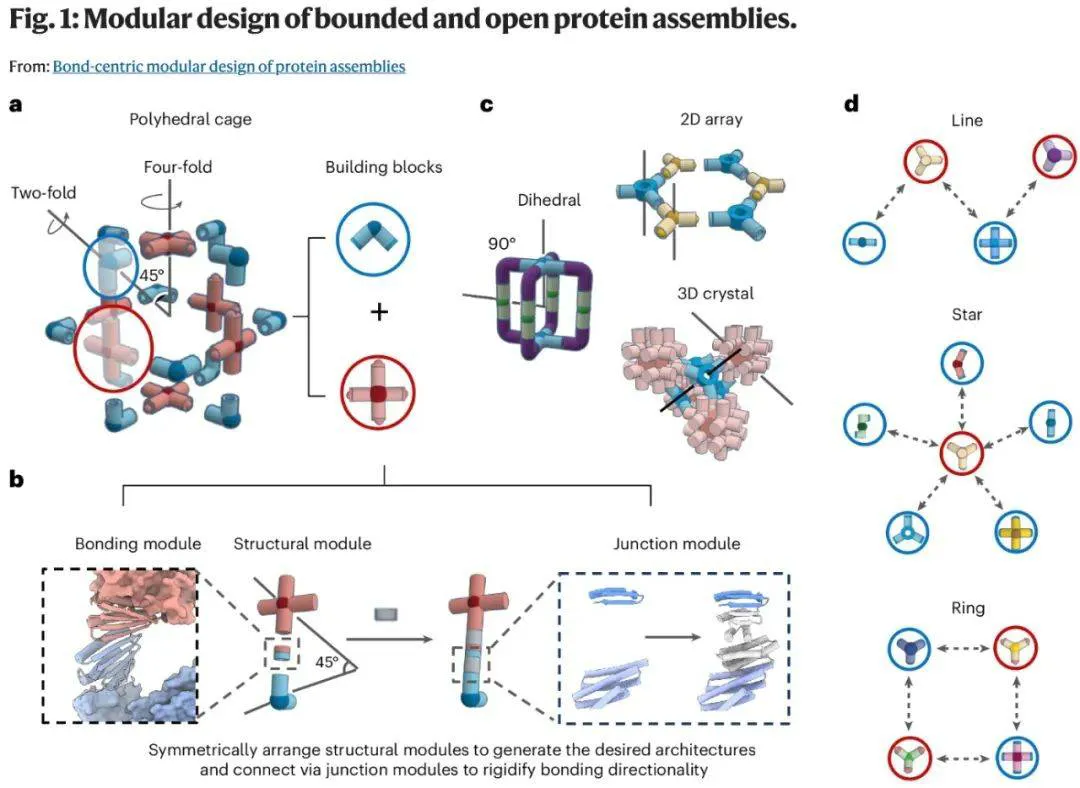
首先,该研究推断:可逆异源二聚体蛋白(如 LHDs)可用作可编程键合模块,应用于具有内部几何匹配特性的寡聚体构建单元。
其次,通过精确调控结构单元主旋转轴之间的特定夹角,该研究实现了不同对称性结构的定向自组装。
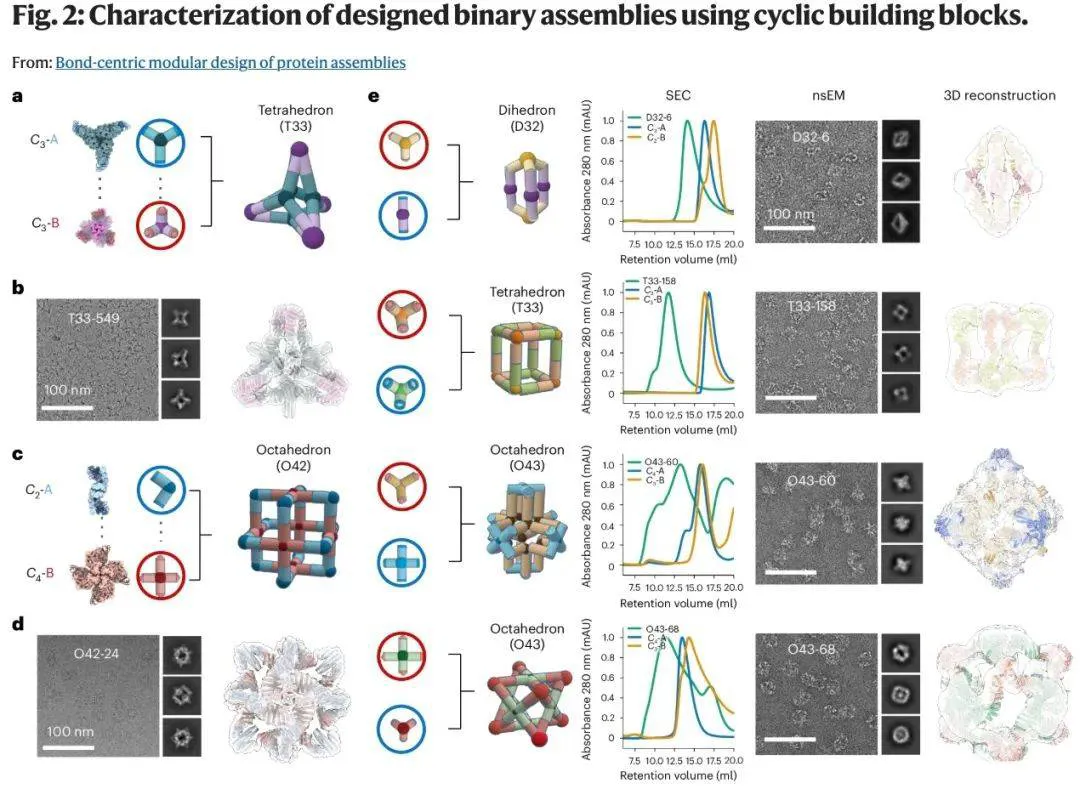
研究的关键在于通过将可逆异源二聚体界面与生成式蛋白质设计相结合,实现了对相互作用方向性的精准调控,从而编程出明确的方向性相互作用。这些标准化的界面与键合几何构型具有高度可预测性,使得仅需少量可重复使用的构建模块即可生成丰富多样的可扩展组装体,极大简化了设计流程。
具体来说,该研究利用 RFdiffusion 进行组装主链设计,用 WORMS 生成主干、ProteinMPNN 进行序列设计,并使用 AlphaFold2 来评估设计的序列是否能够按预期折叠或组装。
精准设计蛋白质纳米材料
相较于传统基于融合的纳米材料设计方法,该研究提出的策略采用标准化可重构界面和定制化构建模块,兼具更高的程序化设计能力与成功率,并能精准调控键合几何构型与相互作用强度,显著拓展了可探索的结构空间。
通过复用已验证组装体的子结构,该研究能够快速构建新型架构,其中笼状结构设计成功率显著提升。如下图所示,单组分多形态的组装特性不仅提高了编码经济性,更为信息存储提供了新途径。
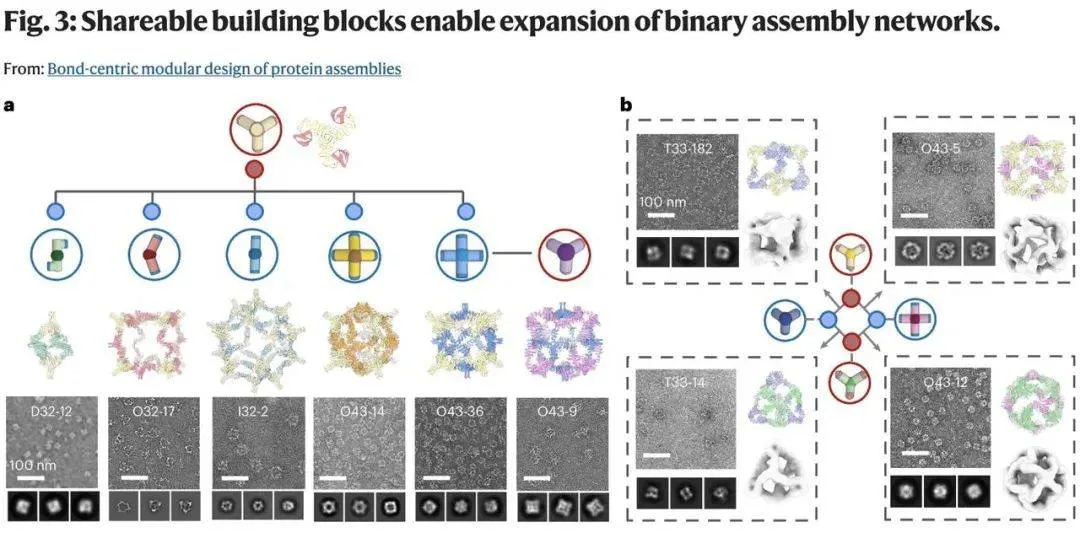
该研究凸显了计算蛋白质设计在开发定制纳米材料方面的潜力,其模块化程度已接近 DNA 纳米技术水平。由于设计的蛋白质可通过遗传编码在多种生命系统中进行表达,因此它们有望作为结构单元、信号单元与控制单元直接整合进活细胞,进而为细胞计算开辟新机遇。
正如标准化零件彻底改变了工业制造,遵循简单规则自组装的标准化蛋白质亚单元,必将推动面向广泛应用的蛋白质组装体开发。



