编辑丨coisini
最近几年,生成式人工智能迅速发展,人们在看到训练机遇的同时也有了风险。由于数据的直接质量影响模型性能,如果训练数据包含有害内容,AI系统可能会继承甚至放大其负面影响。人们开始担心AI会对人类造成威胁。
但如果我们把思路打开,让这种威胁作用在「细菌」上呢?
近日,麻省学院(MIT)生物工程教授吉姆·柯林斯(Jim Collins)领导的研究团队证明:在大量抗菌物质数据集上训练的生成式AI算法,可以帮助科学家构想出数百万种具有繁殖潜力的新型分子,而其中理工部分分子在实验实验中表现出显着效果。
研究论文以《一种从头抗生素设计的生成深度学习方法》为题发表于《Cell》上。

论文地址:https://www.cell.com/cell/abstract/S0092-8674 (25) 00855-4
分子设计
研究团队利用人工智能模型理论可行但尚未存在或生成发现的分子结构,从而探索潜在的药物化合物。
研究团队双管齐下:一方面引导基于抗菌活性化学碎片设计分子的生成式AI算法;另一方面特定的让算法从头生成不可碎片限制的分子结构。
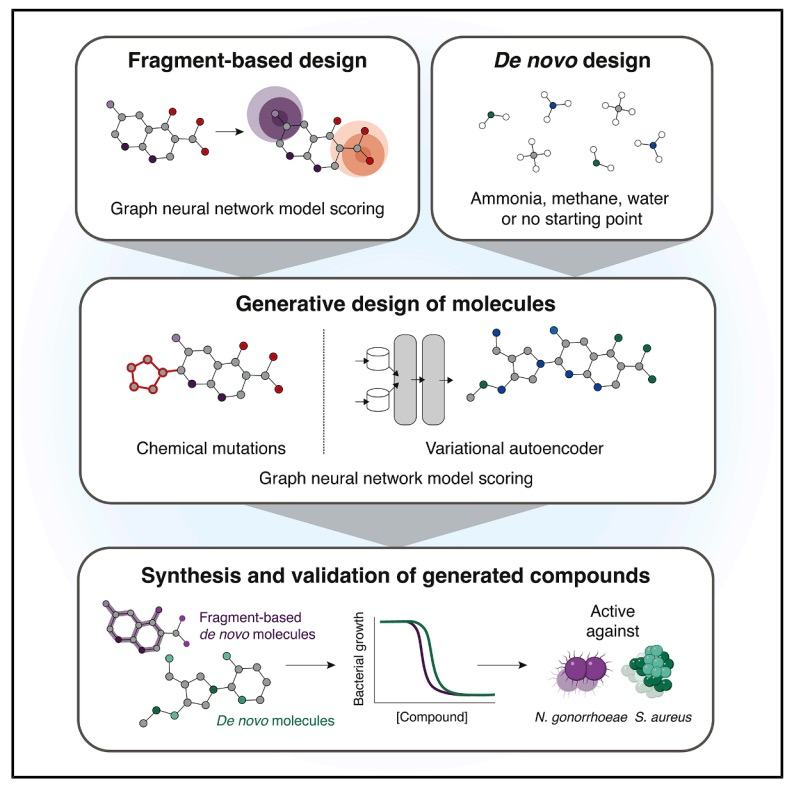
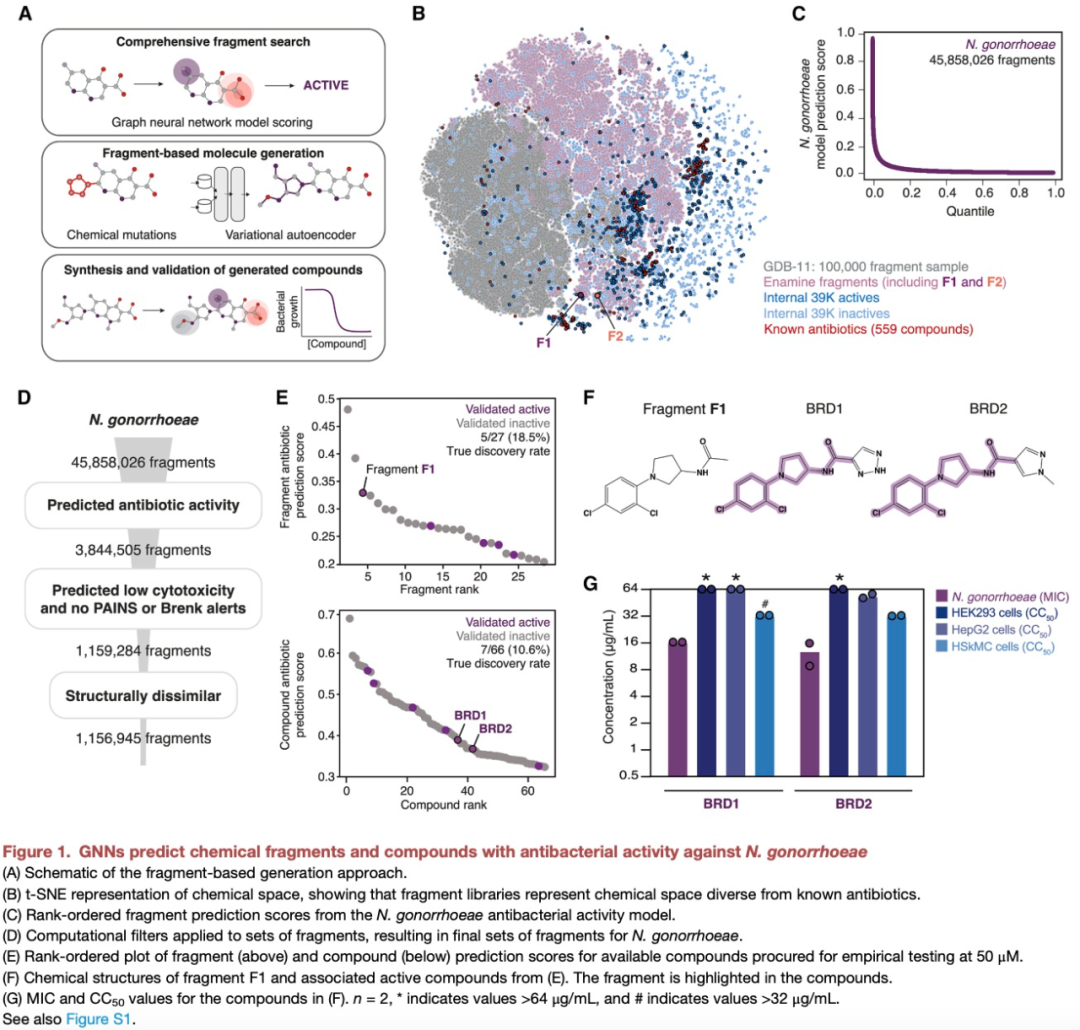
在片段导向方法中,该研究利用GNN对超过4500万个化学片段进行计算机模拟筛选,识别出对淋病奈瑟菌和金葡萄球菌具有惯性抗菌活性预测()的片段。为了将片段扩展为分子,研究团队将其输入两类生成算法——基于化学合理(CReM)的遗传算法和变分自编码器(VAE)。
在从头生成方法中,该研究取消分段输入要求,让CReM和VAE模型训练基于获得的知识自主设计分子。
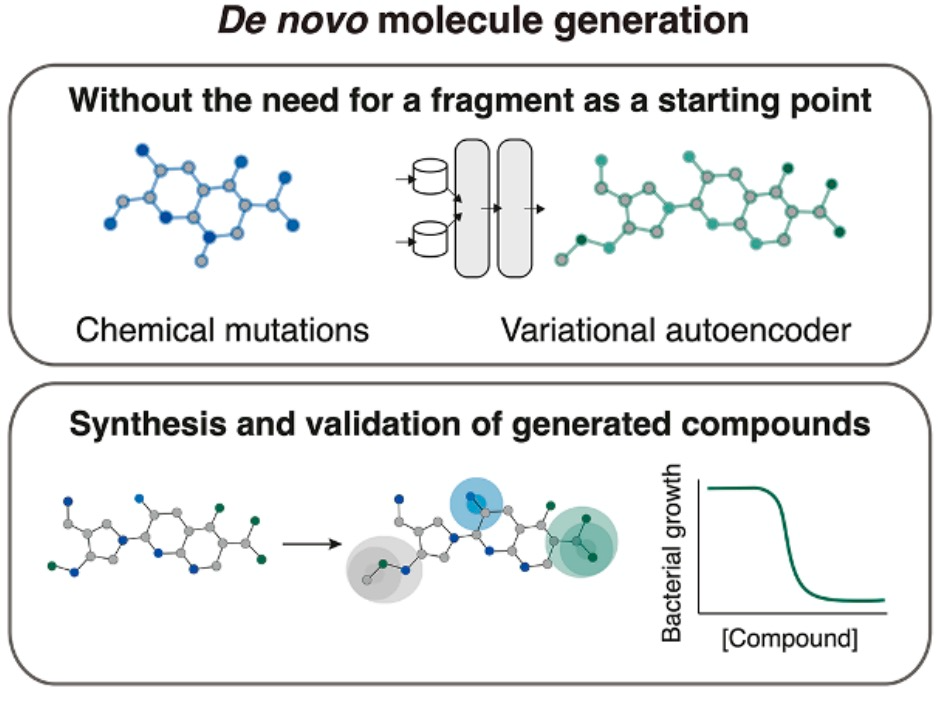
这些模型共同生成了超过 3600 个附件记录的、具有预测抗菌活性的化合物。
合成与验证
尽管分子设计顺利进行,但一系列研究遇到了“巨大极限”:这些预测抗生素大多数无法在实验室实际合成。
经过筛选,研究团队合成了24种化合物,并通过实验验证其中7种具有抗菌活性。
特别是,NG1和DN1两种成分表现出高效和高选择性,其作用机制区别于临床常用抗生素,并在感染模型中验证有效。
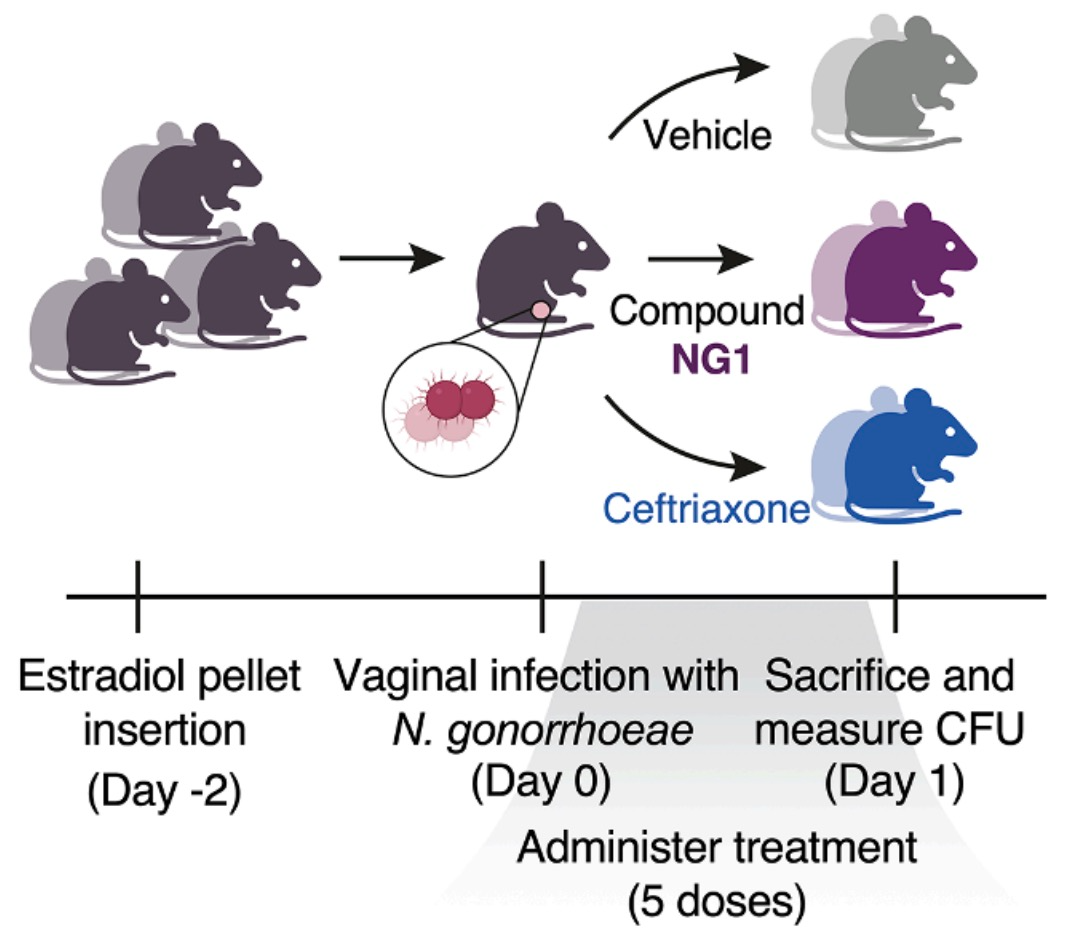
这项研究开辟了一条新路径——生成式AI平台不再从现有化学库中挖掘潜在抗生素,而是从头开始凭空创造出数据库从未记载的全新分子结构。「这其中AI从发现工具转变为设计工具」,Jim Collins表示。
更进一步讲,该研究不仅能够助力无机化合物的研发,还能推动庞大未知化学空间的探索。
参考内容:
https://phys.org/news/2025-08-generative-ai-compounds-drug-resistant.html
https://spectrum.ieee.org/ai-drug-design-mit-antibiotics?utm_source=homepage&utm_medium=hero&utm_campaign=2025-08-14&utm_content=hero3