编辑丨&
在一张桌前有两个人,倘若我们能读懂他们的语言神态,那我们就能推断出下一刻他们会是争吵亦或是握手。
现在,把「人」换成蛋白质,把「话」换成氨基酸序列——蛋白质相互作用是生命过程的核心:信号传导、代谢通路、病毒入侵宿主等,都依赖特定蛋白质之间的物理接触。倘若能读懂其中的相互作用,也就能为接下来的一系列研究铺平道路。
为此,格拉斯哥大学(University of Glasgow)等的研究者提出一种方法,填补现有模型在泛化能力上的缺陷。他们把单蛋白的语言模型扩展为双蛋白的「对话识别器」,让大型蛋白质语言模型学会同时听两条序列,从中判断它们是否会相互作用(PPI)。
相关的研究以「PLM-interact: extending protein language models to predict protein-protein interactions」为题,于 2025 年 10 月 27 日发布在《Nature Communications》。

论文链接:https://www.nature.com/articles/s41467-025-64512-w
一款对接翻译器
在病毒学中,PPIs 尤其重要,因为病毒完全依赖于宿主细胞进行复制,主要通过与宿主蛋白质的特异性相互作用来实现。倘若是能读懂其中的相互作用,就能为开发针对性治疗药物带来更便捷的方法。
计算算法为大规模预测 PPI 提供了一种高效的替代方案。基于大型公共蛋白质序列数据库训练的蛋白质语言模型(PLM)被用于编码序列组成、进化和结构特征,成为表示蛋白质在最新 PPI 预测器中的首选方法。
而该研究团队所提出的 PLM-Interact,通过扩展和微调预训练的 PLM,即 ESM-2,直接建模 PPI。它能把两条蛋白序列拼接入模型,使 Transformer 的注意力能跨蛋白捕捉「配对」信息,而不是先各自编码再拼特征。
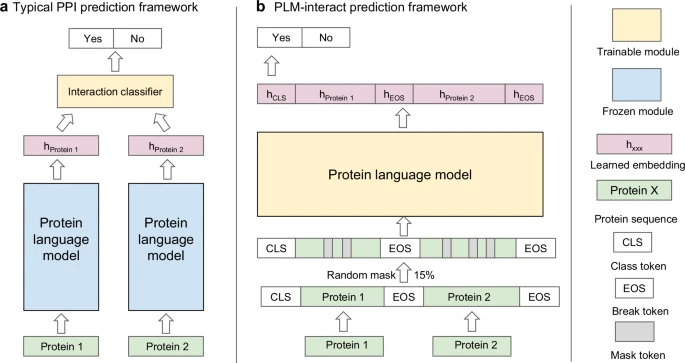
图 1:PLM-interact 与现有 PPI 预测架构的比较。
在掩码语言建模(MLM)任务之外,团队加入l二元分类任务,直接训练模型判断给定的蛋白对是否真实交互。分类损失与 MLM 损失按一定比例(paper 中选择 1:10)混合训练,且模型层被全部微调以适配这一双序列任务。
这些设计让氨基酸残基间的跨蛋白注意力成为可能:模型不再只学习「单句语法」,而能学习两条序列间的「对话格局」。该方法既保留了预训练的结构-进化信息,又把相互作用信号嵌入到语言模型内部。
跨物种提升
为了检验 PLM-interact 的性能,该模型在包含 421,792 对蛋白的训练集(以人类数据为主)进行训练,并与其余六种方法在物种其他物种上进行测试。
相较于六种现有方法,PLM-interact 在 AUPR(精确率-召回曲线下面积)上取得了最佳成绩:在鼠、果蝇和线虫上分别提升约 2%、8%、6%(对比次优方法),在更进化上远的酵母和细菌上也有显著增益。
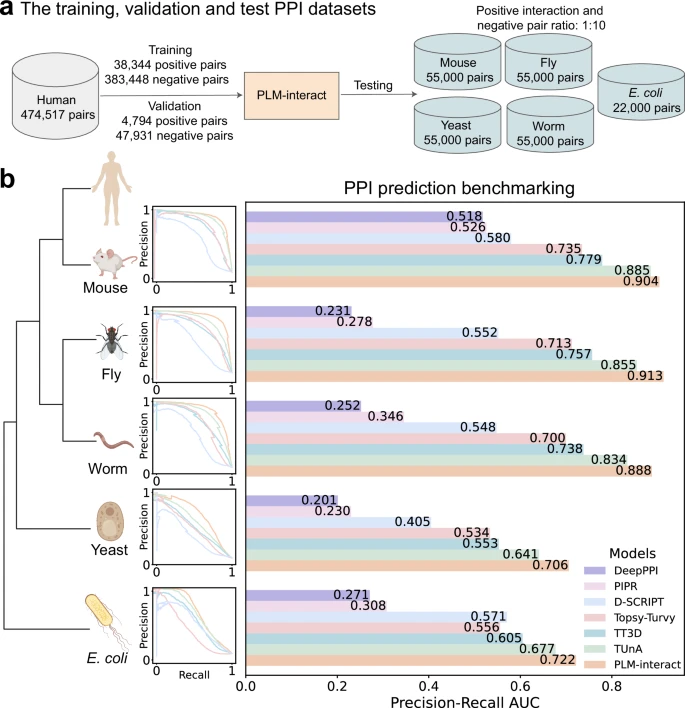
图 2:PLM-interact 与最先进的 PPI 预测模型的基准测试结果。
PLM-interact 的改进归因于其正确识别正 PPI 的能力:它始终将更高的相互作用概率分配给真正的阳性 PPI。相比之下,其他方法在所有保留物种中都给出了较低的相互作用概率估计。
PLM-interact 能够准确预测五个关键的蛋白质相互作用,这些相互作用控制着重要的生物学功能,包括 RNA 聚合和蛋白质运输。值得注意的是,其他蛋白质 AI 工具,包括由谷歌 DeepMind 支持的 AlphaFold3,只能预测五个蛋白质相互作用中的一个。
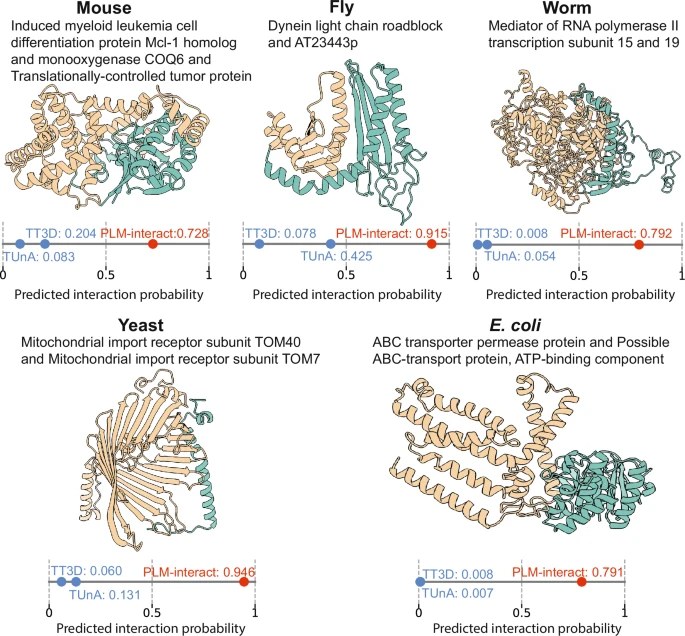
图 3:PLM-interact 正确预测但 TUnA 和 TT3D 未能预测的每个物种的 PPI 示例。
那么,为什么这些举措有用?
把两条序列放进同一个 Transformer,等于是把「对话」上下文纳入模型的注意力机制:模型可以直接把一个蛋白的某个残基与另一个蛋白的某个残基联系起来,形成「跨分子接触」的隐式表示。这比先各自编码再拼接特征更直接,也更容易捕捉到互作所需的互补性。
把分子当语言来读
PLM-interact 展示了一个清晰的理念:把序列看成「句子」,把相互作用看成「对话」,通过模型学习语言级别的跨分子关系,也可以在未见物种上实现高度泛化。
在超过 6.5 亿个独立参数的独立参数的背后,是研究者对开发一个能够以前所未有的规模和精度的,预测蛋白质相互作用的系统所作出的贡献。这项工作是「让超级计算机学会分子语言」的一步,把序列信息转化为可以操作的生物学推断。
相关报道:https://phys.org/news/2025-10-supercomputer-ai-intricate-language-biomolecules.html